SARS-CoV-2 Proteine
antibodies-online bietet eine große Auswahl an rekombinanten Proteinen für die Kitentwicklung und SARS-CoV-2-Forschung, darunter Membranprotein, Nukleokapsidprotein, Spike-Protein, S-Protein-Mutationen, Hüllprotein und Nicht-Strukturproteine. Entdecken Sie unser Produktportfolio.
- Wildtyp, Full-length Cov-2 Spike Protein.
- Für Assay Development.
- Lieferzeit 2-3 Tage.
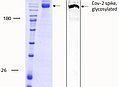
- T19R, L452R, T478K, D614G, P681R, D950N
- Quelle: HEK-293 Cells
- Lieferzeit 2-3 Tage.
- Trimeric, full length Cov-2 Spike Protein.
- Für Assay Entwicklung.
- 2-3 Tage Lieferzeit!

- Spike S1 Protein gelabelt mit His tag.
- Für funktionelle Studien.
- Produkt ABIN6952427

Top Produkte: Trimeric SARS-CoV-2 Spike Protein
- (2)
- (6)
- (5)
- (4)
- (4)
- (4)
*Super Stable Trimer: Prolin-Substitutionen (F817P, A892P, A899P, A942P, K986P, V987P) und Alanin-Substitutionen (R683A und R685A) , um den trimeren Präfusionszustand des SARS-CoV-2 S-Proteins zu stabilisieren bzw. die Furin-Spaltstelle zu eliminieren.
SARS-CoV-2 Spike (S1, S2) Proteine
- (9)
- (6)
- (4)
- (5)
- (4)
- (3)
- (3)
- (3)
- (3)
- (2)
- (1)
- (2)
- (2)
SARS-CoV-2 Nucleocapsid (N) Proteine
SARS-CoV-2 Membran (M) Proteine
SARS-CoV-2 Nichtstrukturproteine (NSP)
- (1)
- (1)
- (1)
- (1)
- (1)
- (1)
- (1)
- (1)
- (1)
Rekombinante SARS-CoV-2-Proteine werden als Antigene für die Antikörperentwicklung, als Capture-Antigene oder als Standards in Assays verwendet. Sie können als Positivkontrolle in Antigen-nachweisenden ELISAs verwendet werden, um positive Ergebnisse genau von falsch-positiv zu unterscheiden, oder als Capture-Antigen für Immunglobulin-ELISAs. Das trimere, Full-Length-SARS-Cov-2-Spike-Protein zum Beispiel eignet sich für die Assay-Entwicklung und ist sehr nützlich bei der Untersuchung neutralisierender Antikörper. Darüber hinaus werden SARS-CoV-2-Proteine für die Medikamentenentwicklung und Studien zur Wiederverwendung von Medikamenten benötigt. In der Wirkstoffforschung sind funktionelle Studien mit aktiven Proteinen unerlässlich, um die hemmende Wirkung der getesteten Substanz zu verifizieren. Sowohl NSPs als auch N-Proteine stehen als potenzielle Targets im Fokus des Interesses; ihre Funktionen und Interaktion mit der Wirtszelle sind entscheidend für die Virusvermehrung und daher von hoher Relevanz für Hemmstrategien. Es hat sich gezeigt, dass das SARS-CoV-2 N-Protein das Komplementsystem beeinflusst, während die SARS-CoV-2 NSPS für die Virusvermehrung verantwortlich sind.
Related Products: full length SARS-Cov-2 Spike protein (ABIN6952670)
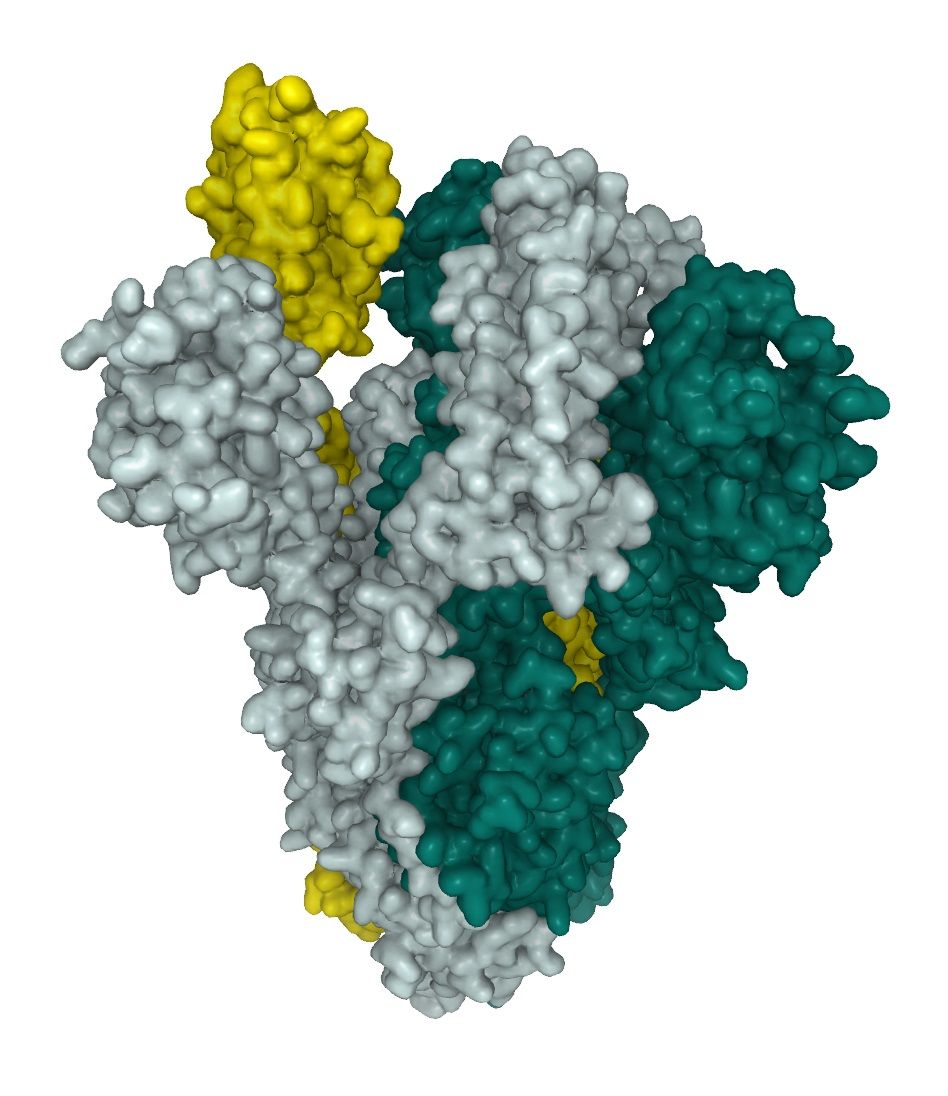
Posttranslationale Modifikationen (PTMs), wie Glykosylierung, modifizieren Proteine als letzten Schritt der Reifung, und fördern Proteinfaltung um die Stabilität zu verbessern. Das Glykosylierungsmuster des SARS-CoV-2-Spike-Proteins liefert ein grundlegendes Verständnis der viralen Struktur und ist wichtig für die Identifizierung von Immunogenen für das Impfstoffdesign, insbesondere im Hinblick auf die sterische Behinderung. Das Spike-Glykoprotein liegt als homotrimeres Fusionsprotein vor. Jedes der Trimere enthält 66 Glykosylierungsstellen für vom Wirt stammende N-gebundene Glykane. Im vorherrschenden Zustand des Trimers befindet sich eine der RBDs in einer "up"-Position, während sich die beiden anderen in einer "down"-Position befinden. Die Interaktion von S-Protein und ACE2 findet nur mit einer RBD in der "up"-Position statt.
SARS-CoV-2 baut mehrheitlich Glykane mit hohem Mannosegehalt sowie komplexe Glykane auf ihren Spike-Proteinen (siehe unten). 1 Dies führt zu einer hochkomplexen Oberflächenstruktur, eine Herausforderung bei der Suche nach Interaktoren und bei der Erzeugung von neutralisierenden Antikörpern (Nabs). Das Full-Length SARS-Cov-2 Spike-Protein (ABIN6952670) wird, wie unsere anderen aktiven SARS-CoV-2-Proteine, in HEK293-Zellen produziert. Das Säugetier-Expressionssystem ist in der Lage, das komplexe Glykosilationsmuster von SARS-CoV-2 nachzuahmen und das homotrimere Fusionsprotein in seinem aktiven "up"-Zustand zu exprimieren (Abb.1). Das Full-Length SARS-Cov-2 Spike-Protein ist daher bei der Untersuchung neutralisierender Antikörper sehr nützlich.
In einem kürzlich erschienenen Glycobiology Artikel führten Shajahan et al. ein ortsspezifisches quantitatives N- und O-gebundenes Glykan-Profiling an rekombinanten SARS-CoV-2 S-Protein-Untereinheiten S1) und SARS-CoV-2 S-Protein-Untereinheiten S2 mittels Glykoproteomik unter Verwendung von hochauflösender LC-MS/MS durch. Das Spike-Protein besteht aus zwei Protein-Untereinheiten (S1 und S2), die zusammen 22 potenzielle N-Glykosylierungsstellen besitzen. Die Gruppe identifizierte 2 unerwartete O-Glykosylierungsstellen an der Rezeptorbindungsdomäne (RBD) der Untereinheit S1.1
Related Products: SARS-CoV-2 Spike Subunit S1 (AA 16-690) protein (His tag) (ABIN6952319) | SARS-CoV-2 Spike Subunit S2 (AA 697-1213) protein (His tag) (ABIN6952319)
Die N-Glykane auf dem S-Protein spielen eine wichtige Rolle bei der korrekten Proteinfaltung und Priming durch Wirtsproteasen. Da die Glykane die Aminosäurereste und andere Epitope vor den Zellen und der Antikörpererkennung schützen können, kann die Glykosylierung das Coronavirus in die Lage versetzen, sowohl der angeborenen als auch der adaptiven Immunantwort zu entgehen.2,3,4 Die Gruppe verwendete rekombinantes SARS-CoV-2 S1 Protein) und SARS-CoV-2 S2 Protein, die in HEK293-Zellen exprimiert wurden, und beobachtete eine partielle Belegung mit N-Glykanen an 17 von 22 N-Glykosylierungsstellen. Zuckerketten mit hohem Mannose Anteil (Man5GlcNAc2) wurden als vorherrschende Struktur an allen Stellen implementiert.5
Thr323 und Ser325 wurden als O-Glykosylierungsstellen auf der S1-Untereinheit des SARS-CoV-2-Spike-Proteins durch hochauflösende massenspektrometrische glykoproteomische Profilierung identifiziert. Die Reste Thr323 und Ser325 befinden sich an der RBD der S1-Untereinheit von SARS-Cov-2, und daher könnte die O-Glykosylierung an dieser Stelle eine entscheidende Rolle bei der viralen Bindung mit hACE2-Rezeptoren spielen.3
Quellen
: "Deducing the N- and O-glycosylation profile of the spike protein of novel coronavirus SARS-CoV-2." in: Glycobiology, Vol. 30, Issue 12, pp. 981-988, (2020) (PubMed).: "Mapping Neutralizing and Immunodominant Sites on the SARS-CoV-2 Spike Receptor-Binding Domain by Structure-Guided High-Resolution Serology." in: Cell, Vol. 183, Issue 4, pp. 1024-1042.e21, (2020) (PubMed).
: "The proximal origin of SARS-CoV-2." in: Nature medicine, Vol. 26, Issue 4, pp. 450-452, (2020) (PubMed).
: "Global aspects of viral glycosylation." in: Glycobiology, Vol. 28, Issue 7, pp. 443-467, (2018) (PubMed).
: "Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV." in: Nature communications, Vol. 11, Issue 1, pp. 1620, (2020) (PubMed).
: "Biochemical characterization of SARS-CoV-2 nucleocapsid protein." in: Biochemical and biophysical research communications, Vol. 527, Issue 3, pp. 618-623, (2020) (PubMed).
: "A SARS-CoV-2-Human Protein-Protein Interaction Map Reveals Drug Targets and Potential Drug-Repurposing." in: bioRxiv : the preprint server for biology, (2020) (PubMed).
: "Immunoinformatic analysis of the SARS-CoV-2 envelope protein as a strategy to assess cross-protection against COVID-19." in: Microbes and infection, Vol. 22, Issue 4-5, pp. 182-187, (2020) (PubMed).
: "Cell entry mechanisms of SARS-CoV-2." in: Proceedings of the National Academy of Sciences of the United States of America, Vol. 117, Issue 21, pp. 11727-11734, (2020) (PubMed).
- Jie Hu et al. The D614G mutation of SARS-CoV-2 spike protein enhances viral infectivity and decreases neutralization sensitivity to individual convalescent sera. bioRxviv (2020).
- Korber B. et al.Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2. bioRxviv (2020). doi.org/10.1101/2020.04.29.069054.
- Lizhou Zhang et al. The D614G mutation in the SARS-CoV-2 spike protein reduces S1 shedding and increases infectivity. bioRxviv (2020). doi.org/10.1101/2020.06.12.148726.
- Junxian Ou et al. Emergence of RBD mutations in circulating SARS-CoV-2 strains enhancing the structural stability and human ACE2 receptor affinity of the spike protein. bioRxiv (2020). doi:10.1101/2020.03.15.991844v4
- Saha, P. et al.Mutations in Spike Protein of SARS-CoV-2 Modulate Receptor Binding, Membrane Fusion and Immunogenicity: An Insight into Viral Tropism and Pathogenesis of COVID-19. chemRxiv (2020). doi:10.26434/chemrxiv.12320567.v1




