COVID-19
Seit dem ersten Auftreten des neuartigen Coronavirus SARS-CoV-2 als Erreger von COVID-19 wurden weltweit fast 5.000.000 Infektionen (Stand: 20. Mai 2020) registriert. Dank weltweiter Forschungsanstrengungen kann der Virus selbst und seine Wirkung auf den menschlichen Wirt Stück für Stück entschlüsselt werden. Wir bieten eine große Auswahl an qualitativ hochwertigen Produkten für die COVID-19-Forschung an.
Antikörper, Kits, Proteine nach Krankheitsphase:
COVID-19 Produkte nach Kategorie:
SARS-CoV-2 Infektion
Die SARS-CoV-2-Infektion wird in erster Linie dann ausgelöst, wenn der Angiotensin-konvertierendes Enzym 2 (ACE2)-Oberflächenrezeptor der Wirtszelle über seine Rezeptorbindungsdomäne (RBD) an das Spike-(S)-Protein des Virus gebunden wird. ACE2 wird auf dem X-Chromosom kodiert, was die höhere COVID-19-Todesfallrate bei Männern erklären könnte. Möglicherweise verleiht das Vorhandensein von zwei verschiedenen ACE2-Allelen einen gewissen Grad an Resistenz.
Die Bindung von SARS-CoV-2 an ACE2 löst das Priming des trimeren S-Proteins an der mehrbasigen S1/S2-Spaltstelle durch die Zelloberflächen-assoziierte Transmembranprotease Serin 2 (TMPRSS2) und in geringerem Maße Cathepsin B und L aus. Die S1-Ektodomäne, die die RBD enthält, bestimmt den zellulären Tropismus und die Anheftung des Virus an seine Zielzelle. Die S2-Endodomäne beherbergt eine Transmembrandomäne und ist am Eintritt des Virus durch Endozytose beteiligt. Es enthält auch eine zweite Protease-Stelle, die furinartige S2'-Spaltungsstelle. Eine Vorspaltung an dieser furinartigen Spaltstelle könnte die höhere Infektiosität von SARS-CoV-2 im Vergleich zu SARS-CoV (dem diese Stelle fehlt) erklären.
Relevante COVID-19 Produkte:
- (8)
- (15)
- (1)
- (4)
- (5)
- (9)
- (8)
- (5)
- (3)
- (4)
- (3)
- (1)
Virale Replikation
Nach dem Eindringen wird das virale Positiv-Sense-ßRNA(+)-Genom freigesetzt. Zwei große polycistronische offene Leserahmen ORF1a und ORF1b am 5'-Ende des Genoms kodieren für 16 nicht-strukturelle Proteine (NSPs), die zwei Replikase-Polyproteine pp1a und pp1b bilden. Nsp3 enthält eine papainähnliche Proteasedomäne (PLpro) und bearbeitet Nsp1-4 von pp1a. Die 3C-ähnliche chymotrypsinähnliche Hauptprotease (Mpro, 3CLpro, Nsp5) von SARS-CoV-2 verdaut die übrigen proteolytischen Spaltstellen.
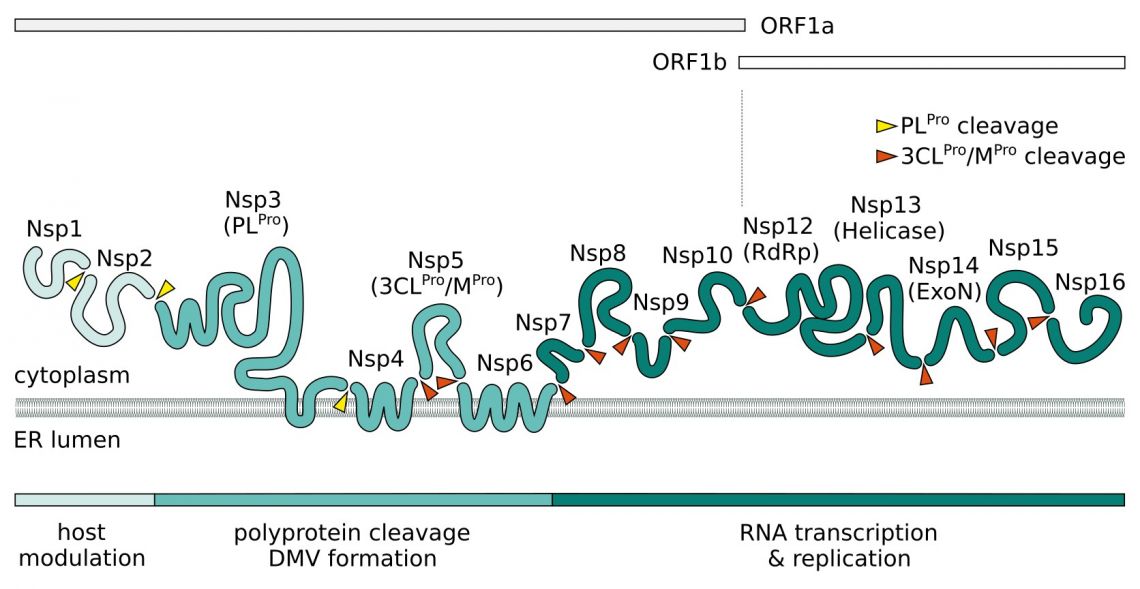
Es wird angenommen, dass Nsp1 und Nsp2 bei der Modulation des Wirts eine Rolle spielen, um eine antivirale Reaktion zu unterdrücken. Ein Komplex, bestehend aus den Transmembranproteinen Nsp3, Nsp4 und Nsp6, induziert die Bildung von Doppelmembranvesikeln (DMV), die die virale Replikation durch membranassoziierte Replikation verbessern und die Autophagie beeinflussen. Zusätzlich zu seinen NSPs rekrutiert SARS-CoV-2 Wirtsproteine zur Bildung eines Replikations- und Transkriptionskomplexes (RTC). Kernkomponente für die Replikation der ssRNA(+) ist die RNA-abhängige RNA-Polymerase (RdRp). Es bildet zusammen mit Nsp7 und Nsp8 den Replikationskomplex. Diese dienen als Primase und erzeugen kurze RNA-Primer für den primerabhängigen RdRp und erhöhen dessen Prozessierbarkeit. Nsp9 hat eine Vorliebe für ssRNA und es wird angenommen, dass es im Replikationskomplex mit Nsp8 interagiert. Nsp13 und Nsp16/Nsp10 haben Helicase/Triphosphatase- bzw. Methyltransferase-Aktivität und kappen die entstehende virale mRNA. Die Exonuklease Nsp14 (ExoN) stattet die Replikationsmaschinerie mit einer Korrekturlesefunktion aus und erhöht so die Genauigkeit der SARS-CoV-2-RNA-Synthese. Das letzte Protein des Replikationskomplexes ist die uridinspezifische Endoribonuklease Nsp15 (EndoU).
Sobald das RTC assembliert ist, wird ein dsRNA-Intermediär aus der genomischen ssRNA(+) synthetisiert. Dieses Zwischenprodukt dient als Vorlage für die Produktion von RNAs in Subgenomlänge (sgRNA) und neuem ssRNA(+)-Genom in voller Länge. Erstere werden in die vier Strukturproteine des Virus (N, E, M und S) und neun akzessorische Proteine, die im 3'-Teil des Genoms kodiert sind, transkribiert. Letzteres wird in nukleokapsidische Phosphoproteine (N-Protein) verpackt und dann von dem Hüllprotein (E-Protein), dem Membranglykoprotein (M-Protein) und dem Spike-Protein (S-Protein) umhüllt, um neue Virionen zu bilden.
Relevante COVID-19 Antikörper:
- (8)
- (15)
- (1)
- (9)
- (8)
- (1)
- (2)
Relevante COVID-19 Proteine:
- (1)
COVID-19
Die Schwere der Erkrankung bei Patienten ist nicht nur auf die Virusinfektion, sondern auch auf die Reaktion des Wirts zurückzuführen. Die Entzündungsreaktion des Wirtes hat einen starken Einfluss auf die Schädigung der Atemwege. In 70% der tödlichen COVID-19-Fälle führt das daraus resultierende akute Atemnotsyndrom (ARDS) direkt zu einem Atemversagen. Die zweithäufigste Ursache für Todesfälle im Zusammenhang mit COVID-19 ist eine unkontrollierte systemische Entzündungsreaktion, die durch eine Überproduktion von Entzündungsmarkern ausgelöst wird. Dieses so genannte Zytokinfreisetzungssyndrom oder Zytokinsturm ist ein wichtiger Mitverursacher des ARDS und des Multiorgan-Dysfunktionssyndroms (MODS), das insbesondere Schäden an Herz, Nieren und Leber verursacht.
SARS-CoV-2-Infektion und Entzündungsreaktion
SARS-CoV-2 vermehrt sich hauptsächlich in den unteren Atemwegen, wo es Lungenentzündung und ARDS verursacht. Während die strukturellen und nicht-strukturellen Proteine des Virus hauptsächlich für den Aufbau der Virion- bzw. Virusreplikation zuständig sind, sind zumindest einige der Mitglieder der dritten Gruppe von SARS-CoV-2-Proteinen, die neun akzessorischen Faktoren (Orf3a-10), an der Progression von COVID-19 beteiligt.
SARS-CoV-2 Orf3a induziert Apoptose in Zelllinienmodellen und soll NF-kB und das NLRP3-Inflammasom aktivieren, das an der Pyroptose, einer hochgradig entzündlichen Form der Apoptose, beteiligt ist. Es konnte gezeigt werden, dass Orf8b ER-Stress induziert und auch das NLRP3-Inflammasom aktiviert. Dies deutet darauf hin, dass eine Virusinfektion der Epithelzellen der Atemwege zu Pyroptose führt, einer hochgradig entzündlichen Form des programmierten Zelltods. Die Pyroptose geht typischerweise mit der Freisetzung proinflammatorischer Zytokine einher, was zur Rekrutierung zusätzlicher Immunzellen und zu einer weiteren Verstärkung der Immunantwort führt. Orf3a induziert auch die Sekretion des Pyroptose-Markers IL-1 beta. Orf7a könnte auch über seine Rolle bei der virusinduzierten Apoptose eine Rolle in der Pathogenese spielen.
Das Ausmaß der Gewebeschädigung in den unteren Atemwegen kann in den frühen Stadien von COVID-19 überwacht werden, wobei die Werte des C-reaktiven Proteins (CRP) als Indikator für die Schwere der Erkrankung dienen.
Die Zerstörung der Alveolarepithelzellen setzt schädigungsassoziierte molekulare Muster (DAMPs) und pathogenassoziierte molekulare Muster (PAMPs) frei, die von Mustererkennungsrezeptoren (PRRs) auf Alveolarepithelzellen und Makrophagen erkannt werden. Die primären PRRs für virale RNAs sind Mitglieder der Familie der RIG-I-ähnlichen Rezeptoren (RIG-I-like receptor, RLR). Bei der Bindung an virale RNA löst eine Konformationsänderung der RLRs die Aggregation von MAVS und die Bildung des MAVS-Signalosoms aus. Das MAVS-Signalosom löst die IRF3/7-Dimerisierung aus und aktiviert den NF-κB-Signalweg, was zur Produktion von Typ-I-IFNs, den wichtigsten antiviralen Zytokinen, und den proinflammatorischen Zytokinen IL-6, IFN-gamma, CD46 und CXCL10 führt. Die koordinierte Sekretion von proinflammatorischen Zytokinen und Chemokinen führt zur Rekrutierung von Immunzellen, insbesondere CD4+ T-Helferzellen (TH1), CD8+ zytotoxischen T-Zellen und Monozyten, um die Abwehr der Virusinfektion zu stärken.
Als Typ-I-IFN-Antagonist hemmt SARS-Cov-2 Orf6 die IFN-Reaktion. Orf9b zielt auf die Degradation des MAVS-Signalosoms und begrenzt daher die Interferonreaktionen der Wirtszelle. Orf9c interagiert mit der mitochondrialen Elektronentransportkette, die an der TLR/IL-1-Signalisierung und der Regulation der Entzündung beteiligt ist. Nsp1 unterdrückt die IFN-Induktion und erhöht die CCL5-Produktion und trägt so zu Entzündungsprozessen bei.
SARS-CoV-2 Interferon Antagonismus: Proteine & Antikörper
SARS-CoV-2 Zytokinsturm & ELISA Kits
Relevante Antikörper: CRP, NFKB1, NFKB2, NFkB cRel, p65, RELB
- (2)
- (7)
- (1)
- (60)
- (5)
- (1)
- (1)
- (1)
- (2)
- (4)
- (5)
Related ORF Proteins:
Unsere Produkte sind nur für Forschungszwecke bestimmt, nicht für diagnostische, therapeutische oder andere Zwecke.
Weiterführende Informationen und Produkte
SARS-CoV-2 Neutralizing Antibodies based on CR3022
SARS-CoV Protein Interactome / poster and products
SARS-CoV-2 RT-PCR Kits and qPCR Kits
SARS-CoV-2 Life Cycle: Stages and Inhibition Targets
Coronavirus Expression Vectors and Cloning Vectors at genomics-online.com.
Referenzen
- Banerjee, A.K. et al. SARS-CoV-2 Disrupts Splicing, Translation,and Protein Trafficking to Suppress Host Defenses. Cell. doi: 10.1016/j.cell.2020.10.004. Link to Pubmed
- Blanco-Melo, D. et al. Imbalanced Host Response to SARS-CoV-2 Drives Development of COVID-19. Cell. 181, 1036 (2020). Link to Pubmed
- Coutard, B. et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. 176, 104742 (2020). Link to Pubmed
- Daly, J. et al. Neuropilin-1 is a host factor for SARS-CoV-2 infection. Science (2020). doi: 10.1126/science.abd3072 Link to Pubmed
- de Wilde, A. H. et al. Host Factors in Coronavirus Replication. in Roles of Host Gene and Non-coding RNA Expression in Virus Infection. Current Topics in Microbiology and Immunology (eds. Tripp, R. A. & Tompkins, S. M.) 1–42 (Springer International Publishing, 2018). doi:10.1007/82_2017_25 Link to Pubmed
- Diao, B. et al. Reduction and Functional Exhaustion of T Cells in Patients With Coronavirus Disease 2019 (COVID-19). Front. Immunol. 11, 827 (2020). Link to frontiersin.org
- Gao, T. et al. Highly pathogenic coronavirus N protein aggravates lung injury by MASP-2- mediated complement over-activation. medRxiv (2020). doi:10.1101/2020.03.29.20041962 Link to medRxiv
- Gao, Y. et al. Structure of RNA-dependent RNA polymerase from 2019-nCoV, a major antiviral drug target. bioRxiv 2020.03.16.993386 (2020). doi:10.1101/2020.03.16.993386 Link to bioRxiv
- Gordon, D. E. et al. A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature (2020). doi:10.1038/s41586-020-2286-9 Link to nature.com
- Hoffmann, Markus; Kleine-Weber, H. et al. SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor. Cell 181, 1–10 (2020). Link to Pubmed
- Pachetti, M. et al. Emerging SARS‑CoV‑2 mutation hot spots include a novel RNA-dependent- RNA polymerase variant. J. Transl. Med. 18, 1–9 (2020). Link to Pubmed
- Shi, Y. et al. COVID-19 infection: the perspectives on immune responses. Cell Death Differ. (2020). doi:10.1038/s41418-020-0530-3 Link to Pubmed
- Tan, C. et al. C‐reactive protein correlates with computed tomographic findings and predicts severe COVID‐19 early. J. Med. Virol. jmv.25871 (2020). doi:10.1002/jmv.25871 Link to Pubmed
- Tay, M. et al. The trinity of COVID-19: immunity, inflammation and intervention. Nat. Rev. Immunol. 1–12 (2020). doi:10.1038/s41577-020-0311-8 Link to Pubmed
- van den Berg, D. H., te Velde, A. A. Severe COVID-19: NLRP3 Inflammasome Dysregulated. Frontiers in Immunology (2020). doi: 10.3389/fimmu.2020.01580 Link to Pubmed
- Wilk, A. et al. A single-cell atlas of the peripheral immune response in patients with severe COVID-19. Nat.Med. (2020). doi:10.1101/2020.04.17.20069930 Link to Pubmed
- Wrapp, D. et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 367, 1260–1263 (2020). Link to Pubmed
- Yap, J. K. Y. et al. Inflammasomes and Pyroptosis as Therapeutic Targets for COVID-19. Journal of Immunology 205, 307-312(2020). Link to Pubmed
- Zhang, L. et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. Science. 368, 409 LP – 412 (2020). Link to Pubmed
- Zhang, D. et al. COVID-19 infection induces readily detectable morphological and inflammation-related phenotypic changes in peripheral blood monocytes, the severity of which correlate with patient outcome. medRxiv (2020). doi:10.1101/2020.03.24.20042655 Link to medRxiv
- Ziegler-Heitbrock, L. The CD14+ CD16+ blood monocytes: their role in infection and inflammation. J. Leukoc. Biol. 81, 584–592 (2007). Link to Pubmed




