CUT&RUN
CUT&RUN steht für "cleavage under targets and release using nuclease". Die vom Henikoff-Labor entwickelte Technik bietet einen neuen Ansatz für die Verfolgung der Epigenetik. CUT&RUN führt einige wichtige Änderungen ein, um die mit der ChIP-Seq verbundenen Mängel zu beseitigen. Es ist einfach durchzuführen und von Natur aus robust, mit extrem niedrigem Hintergrund, der nur ~1/10 der Sequenzierungstiefe von ChIP erfordert. CUT&RUN ist kostengünstig für die Erstellung von Transkriptionsfaktor- und Chromatinprofilen.
CUT&RUN Antibodies
Eine Auswahl von Antikörpern, die für die Verwendung in CUT&RUN-Tests validiert sind.
CUT&RUN Sets
CUT&RUN leicht gemacht - mit unseren Sets & Komponenten
CUT&RUN ConA Beads
Magnetic ConA Beads für die Nutzung in CUT&RUN Assays
CUT&RUN - Besser als ChIP-seq!
CUT&RUN-Workflow: Extraktion ohne Fragmentierung
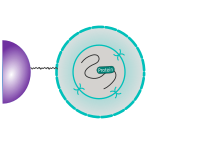
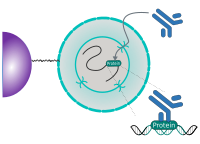
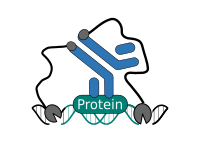
CUT&RUN wird in situ an immobilisierten, intakten Zellen ohne Crosslinking durchgeführt. Die DNA-Fragmentierung erfolgt durch Mikroko nuclease, die mit Protein A und/oder Protein G (pAG-MNase) fusioniert ist. Das Fusionsprotein wird über die Bindung der Protein A/G-Domäne an die Fc-Region eines an das Ziel gebundenen Antikörpers zum gewünschten Ziel geführt. Die unter dem Ziel befindliche DNA wird anschließend geschnitten und freigesetzt, wobei der pAG-MNase-Antikörper-Chromatin-Komplex aus der Zelle diffundieren kann. Die DNA-Spaltprodukte werden extrahiert und anschließend mittels Next Generation Sequencing (NGS) weiterverarbeitet.
Das Standard-CUT&RUN-Protokoll ist in erster Linie für das Profiling von Histonproteinen und Transkriptionsfaktoren vorgesehen, die an Chromatin binden. Der zuverlässige Nachweis seltener Transkriptionsfaktoren, transienter Interaktionen oder von Proteinen in Komplexen, die nicht direkt mit DNA assoziiert sind, hat sich bei Verwendung von CUT&RUN an ungekoppelten Proben als schwierig erwiesen. Die Erhaltung von Protein-Protein- und DNA-Protein-Interaktionen durch Crosslinker kann hingegen Artefakte verursachen. Die folgende Abbildung zeigt das Standard-CUT&RUN-Protokoll:
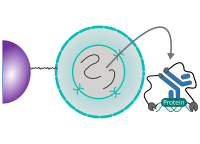
CUT&RUN Low Volume and Urea (CUT&RUN Lov-U) ist eine Modifikation des ursprünglichen Verfahrens, die auf schwer charakterisierbare Interaktionen in situ abzielt und dabei auf Crosslinking verzichtet. Die initiale nukleäre Extraktion minimiert die Sequestrierung des primären Antikörpers und der pAG-MNase durch nicht-chromatin-gebundene Ziele im Zytosol. Die Verwendung niedriger Volumina während des gesamten Protokolls erleichtert die parallele Verarbeitung von Proben, was die Skalierbarkeit und Reproduzierbarkeit verbessert. Schließlich wird Harnstoff als chaotropes Mittel eingesetzt, um Proben in situ zu denaturieren und CUT&RUN-Produkte freizusetzen, gefolgt von einer DNA-Reinigung an Beads.
Ein Nachteil von CUT&RUN ist die Notwendigkeit des Endpolierens und der Ligation von Sequenzieradaptern vor der Herstellung einer Sequenzierbibliothek. Die Kombination des CUT&RUN-Protokolls mit einer Tagmentierung durch eine hyperaktive Tn5-Transposase führte zur Entwicklung der CUT&Tag-Methode. Zellen werden dabei mittels magnetischer Concanavalin-A-Beads immobilisiert und reversibel durch Digitonin permeabilisiert. Anstelle des gerichteten Nuklease-Schnitts wird die DNA jedoch durch einen pA/G-Tn5, der mit Sequenzieradapter-Duplexen beladen ist, fragmentiert. Die Sequenzieradapter werden direkt während der Tagmentierung an die DNA-Fragmente angefügt. Eine weitere DNA-Endbearbeitung ist nicht erforderlich, und die Fragmente können unmittelbar zur Sequenzierbibliothek weiterverarbeitet werden. Zusätzlich ist CUT&Tag aufgrund der Fragmentierung an intakter doppelsträngiger DNA weniger anfällig für Schäden an endogener DNA als CUT&RUN.
Erfahren Sie mehr über CUT&Tag: Workflow, Vorteile, Produkte
CUT&RUN Antibodies
Eine Auswahl von Antikörpern, die für die Verwendung in CUT&RUN-Tests validiert sind.
CUT&RUN Sets
CUT&RUN leicht gemacht - mit unseren Sets & Komponenten
CUT&RUN ConA Beads
Magnetic ConA Beads für die Nutzung in CUT&RUN Assays
Holen Sie sich jetzt unser kostenloses CUT&RUN- und CUT&Tag-Handbuch!
Mehr sehenZielantigene bei CUT&RUN und CUT&Tag
Histonmodifikationen wie das repressive H3K27me3 oder das mit aktiven Promotoren assoziierte H3K4me3 sind häufig verwendete Zielantigene sowohl für CUT&RUN als auch für CUT&Tag. Weitere Zielantigene umfassen Transkriptionsregulatoren wie die Transkriptionsfaktoren CTCF oder SOX2, Chromatin-Modifikatoren wie HDAC2 oder Proteine, die am Nukleinsäurestoffwechsel beteiligt sind, wie Pol II. Zu den nicht-proteinhaltigen Zielen gehören G-Quadruplexe und m6A. Tags wie beispielsweise FLAG, HA oder GFP sind Optionen für rekombinant exprimierte Zielproteine.

