CUT&RUN
CUT&RUN steht für "cleavage under targets and release using nuclease". Die vom Henikoff-Labor entwickelte Technik bietet einen neuen Ansatz für die Verfolgung der Epigenetik. CUT&RUN führt einige wichtige Änderungen ein, um die mit der ChIP-Seq verbundenen Mängel zu beseitigen. Es ist einfach durchzuführen und von Natur aus robust, mit extrem niedrigem Hintergrund, der nur ~1/10 der Sequenzierungstiefe von ChIP erfordert. CUT&RUN ist kostengünstig für die Erstellung von Transkriptionsfaktor- und Chromatinprofilen.
CUT&RUN Antibodies
Eine Auswahl von Antikörpern, die für die Verwendung in CUT&RUN-Tests validiert sind.
CUT&RUN Sets
CUT&RUN leicht gemacht - mit unseren Sets & Komponenten
CUT&RUN ConA Beads
Magnetic ConA Beads für die Nutzung in CUT&RUN Assays
CUT&RUN - Besser als ChIP-seq!
Starten Sie jetzt mit CUT&RUN
Antibodies-online bietet ein umfassendes Angebot an Produkten für CUT&RUN: CUT&RUN-Sets, für CUT&RUN validierte Antikörper, pAG-MNase, magnetische Concanavalin A-Beads, Positiv- und Negativkontroll-Antikörper und mehr. Unser Sekundärantikörper ABIN101961 wurde in verschiedenen Originalarbeiten für CUT&RUN von S. Henikoff verwendet. Entdecken Sie unsere Produktpalette.
CUT&RUN Antibodies
Eine Auswahl von Antikörpern, die für die Verwendung in CUT&RUN-Tests validiert sind.
CUT&RUN Sets
CUT&RUN leicht gemacht - mit unseren Sets & Komponenten
CUT&RUN ConA Beads
Magnetic ConA Beads für die Nutzung in CUT&RUN Assays
Holen Sie sich jetzt unser kostenloses CUT&RUN- und CUT&Tag-Handbuch!
Mehr sehenWarum sollten Sie sich für CUT&RUN-Produkte von antibodies-online.com entscheiden?
Wir von antibodies-online haben den großen Nutzen der CUT&RUN-Technologie schon sehr früh erkannt und uns besonders intensiv damit beschäftigt. Wir arbeiten intensiv an Produkten, die diese innovative Methode noch effizienter und einfacher machen. Unsere Erkenntnisse stellen wir regelmäßig in unserem eigenen CUT&RUN-Protokoll sowie in Webinaren und Broschüren zur Verfügung.
Unsere CUT&RUN-Antikörper, -Sets und -Komponenten bieten Ihnen höchste Qualität und Zuverlässigkeit. Bei Fragen wenden Sie sich bitte an unseren CUT&RUN-Experten Dr. Stefan Pellenz.
- Umfangreich validierte Produkte
- Komponenten präzise aufeinander abgestimmt
- Mehr und mehr für CUT&RUN validierte Antikörper
- Wissenschaftlicher Support bei Fragen und Problemen
Die Bedeutung der Antikörperauswahl

antibodies-online unterstützt die Validierung von Antikörpern für CUT&RUN. Gemeinsam mit Kunden und Herstellern führen wir derzeit mehr als 50 Validierungsprojekte durch. Und fast wöchentlich kommen neue hinzu. Sie können an unserer unabhängigen Validierungsinitiative (IVI) teilnehmen. Schlagen Sie ein Validierungsexperiment vor und führen Sie es durch (CUT&RUN) und erhalten Sie die volle Kostenerstattung für den validierten Antikörper. Kontaktieren Sie uns unverbindlich per E-Mail!
CUT&RUN: Häufig gestellte Fragen
- Was sind die Vor- und Nachteile von CUT&RUN und CUT&Tag im Vergleich zu ChIP-seq? Wie entscheide ich mich für eine der beiden Methoden?
- Warum ist die DNA-Ausbeute so gering?
- Wie wähle ich einen primären Antikörper für CUT&RUN oder CUT&Tag?
- Warum brauche ich einen negativen Kontrollantikörper für CUT&RUN? Warum kann ich nicht einfach eine Kontrolle ohne Antikörper verwenden?
- Kann ich die Antikörper-Negativkontrolle für CUT&RUN durch eine Knock-out- (oder Knock-down-) Variante meines Proteins ersetzen?
- Muss ich für CUT&RUN und CUT&Tag einen sekundären Antikörper verwenden?
- Einer meiner Antikörper ist von der Maus. Hat Ihre pAG-MNase eine gute Affinität zu Maus-Antikörpern, oder raten Sie zur Verwendung eines Kaninchen-Anti-Maus-Sekundärantikörpers?
- Sollte ich heterologe Spike-in-DNA für die Quantifizierung einschließen?
- Ist es möglich, die Zellen vor der Immobilisierung zu fixieren?
- Ist es möglich, die CUT&RUN-Antikörper-Online-Produktsets mit pflanzlichen Gewebeproben zu verwenden?
- Ist es möglich, CUT&RUN an RNA-bindende Proteine anzupassen?
- Kann ich anstelle des Proteinase-K-Verdaus die Proteine in den CUT&RUN-Produktkomplexen durch Hitze denaturieren?
- Was ist für die DNA-Extraktion vor der Bibliotheksvorbereitung vorzuziehen: Extraktion mit Phenol-Chloroform oder Affinitätsreinigung mit einer Säule?
- Ist es möglich, die CUT&RUN-Bibliotheken im Single-End- statt im Paired-End-Verfahren zu sequenzieren?
Entdecken Sie unser CUT&RUN / CUT&Tag Handbuch!
AnsehenWas sind die Vor- und Nachteile von CUT&RUN und CUT&Tag im Vergleich zu ChIP-seq? Wie wähle ich zwischen beiden Methoden?
Die Vorteile von CUT&RUN und CUT&Tag im Vergleich zu ChIP-seq sind ein besseres Signal-Rausch-Verhältnis, eine höhere Empfindlichkeit, ein größerer dynamischer Bereich, ein geringerer Bedarf an Sequenzier-Reads und eine geringere Zellzahl.
CUT&Tag hat den Vorteil, dass die Sequenzierprimer an die gespaltenen DNA-Fragmente angehängt werden und weniger Schritte zur Bibliotheksvorbereitung erforderlich sind als bei CUT&RUN. Ein zusätzliches Annealing ist nicht erforderlich. Die Methode eignet sich besonders gut für nukleosomale und fest gebundene Proteine. Sie wurde vom Henikoff-Labor auch zu einem Protokoll gestrafft, bei dem der gesamte Prozess in einem Röhrchen abläuft, und es sind automatisierbare Varianten mit hohem Durchsatz verfügbar.
CUT&RUN hingegen ist für Transkriptionsfaktoren und andere weniger fest gebundene DNA-bindende Proteine vorzuziehen, die empfindlich auf die höhere Salzkonzentration in CUT&Tag reagieren, die notwendig ist, um eine Off-Target-Markierung von zugänglichem Chromatin durch Tn5 zu verhindern. Darüber hinaus ist die räumliche Auflösung des MNase-Verdaus höher als die der Tagmentierung, was einen deutlicheren Fußabdruck des gewünschten Proteins ermöglicht.
Warum ist die DNA-Ausbeute so gering?
CUT&RUN und CUT&Tag werden mit geringen Zellzahlen durchgeführt, und das Hintergrundsignal ist deutlich geringer als z. B. bei ChIP. Dies kann zuverlässige Messungen der DNA-Konzentration mit einem fluorometrischen Assay oder durch Kapillarelektrophorese schwierig machen.
Um den Erfolg der CUT&RUN- und CUT&Tag-Methoden zu beurteilen, wird empfohlen, eine Reaktion mit einem Antikörper gegen eine häufige Histonmodifikation wie h4K27me (ABIN6923144) oder h4K4me3 (ABIN2668472) als Positivkontrolle durchzuführen. DNA-Fragmente, die mit einem solchen Antikörper präpariert wurden, können durch Kapillarelektrophorese auf einem Bioanalyzer oder Tapestation oder fluorometrisch auf einem Qubit- oder Nanodrop-Fluorometer gemessen werden.
Wie wähle ich einen primären Antikörper für CUT&RUN oder CUT&Tag aus?
Antikörper, die für ChIP-seq empfohlen werden, funktionieren nicht unbedingt in CUT&RUN in CUT&Tag. Im Gegensatz zu ChIP-seq liegt das Antigen im Allgemeinen in seinem nativen Zustand ohne zusätzliche Fixierung vor. Sofern ein Antikörper nicht bereits auf CUT&RUN/Tag getestet wurde, ist eine Empfehlung für eine Methode hilfreich, bei der das Antigen voraussichtlich in nativem Zustand vorliegt, z. B. Immunfluoreszenz. Sofern nicht anders angegeben, ist die empfohlene Verdünnung für die Immunfluoreszenz auch ein guter Ausgangspunkt für die Konzentration des Antikörpers in CUT&RUN/Tag.
Warum brauche ich einen negativen Kontrollantikörper für CUT&RUN? Warum nicht einfach eine Kontrolle ohne Antikörper verwenden?
Die für CUT&RUN verwendete MNase ist eine Endo- und Exonuklease, die unspezifisch an ungeschützte DNA in schwer zugänglichen Bereichen bindet und diese spaltet, z. B. in Regionen um regulatorische Elemente. Freie MNase schneidet bevorzugt DNA in diesen schwer zugänglichen Regionen, was zu falsch positiven Ergebnissen führen und das Hintergrundsignal im Allgemeinen erhöhen kann.
Um diesen unerwünschten Effekt ungebundener MNase zu vermeiden, wird das Chromatin vor der Zugabe von pAG-MNase (ABIN6950951) zu den Proben zufällig mit dem CUT&RUN-Antikörper gegen Kaninchen-IgG als Negativkontrolle beschichtet (ABIN101961). pAG-MNase wird dann über seinen Protein-A- oder Protein-G-Teil an das Fc-Fragment des Antikörpers gebunden, und die Hintergrund-DNA-Fragmentierung wird durch die zufällige Antikörperbindung bestimmt und nicht durch den Nuklease-Verdau der schwer zugänglichen DNA-Regionen.
Kann ich die Antikörper-Negativkontrolle für CUT&RUN durch einen Knock-out (oder Knock-down) meines Proteins ersetzen?
Beide Kontrollen sind nützlich, betreffen aber unterschiedliche Aspekte des Experiments und sind daher nicht austauschbar.
Der CUT&RUN-Antikörper gegen Kaninchen-IgG vom Meerschweinchen (ABIN101961) wird als negative Kontrolle verwendet, um einen Referenzhintergrund für das Peak-Calling zu erhalten. Dies ist notwendig, da das Hintergrundsignal in CUT&RUN-Proben im Vergleich zu ChIP-seq-Proben sehr gering ist. Die ko- (oder kd-) Kontrolle hingegen vermittelt den Eindruck einer unspezifischen Bindung des gegen das betreffende Protein gerichteten Antikörpers an andere Proteine. Sie ist nützlich, um die Identifizierung von falsch positiven Signalen zu vermeiden.
Benötige ich einen sekundären Antikörper für CUT&RUN und CUT&Tag?
Je nach Wirtsspezies und Isotyp des Antikörpers und des MNase-Fusionsproteins Protein A und/oder Protein G kann ein sekundärer Antikörper für die MNase-Bindung erforderlich sein. Protein A hat eine gute, hohe Affinität zu allen Kaninchen-IgG-Antikörpern, aber eine geringe Affinität zu Antikörpern des IgG-Isotyps von Ratte, Ziege und Schaf sowie zu bestimmten IgG-Antikörper-Unterklassen der Maus, insbesondere IgG1. Protein G hingegen bindet gut an die Fc-Region von Maus-, Ziegen-, Schaf- und den meisten Ratten-IgG. Seine Affinität zu Kaninchen-IgG ist jedoch geringer als die von Protein A. Bei der Verwendung von pAG-MNase, die mit dem verbesserten CUT&RUN-Protokoll eingeführt wurde, ist es daher im Allgemeinen nicht erforderlich, einen sekundären Antikörper zu verwenden. Die Verwendung der pA-MNase des ursprünglichen Protokolls kann jedoch die Verwendung eines in Kaninchen gezüchteten sekundären Antikörpers erfordern, um eine effiziente Bindung des Fusionsproteins an den Antikörper zu gewährleisten.
Für CUT&Tag wird ein sekundärer Antikörper empfohlen, um die lokale Konzentration der Fc-Fragment-Bindungsstelle in der Nähe der beabsichtigten Umsetzungsstelle um das interessierende Antigen zu erhöhen. Dieser Schritt ist notwendig, um das spezifische Signal zu erhöhen.
Einer meiner Antikörper ist von der Maus. Hat Ihre pAG-MNase eine gute Affinität für Maus-Antikörper, oder raten Sie zur Verwendung eines sekundären Kaninchen-Anti-Maus-Antikörpers?
Die pAG-MNase wird gut mit Ihrem Maus-Antikörper funktionieren. Der Zusatz von Protein G zur MNase dient in erster Linie dazu, die Verwendung von monoklonalen Maus-IgG1-Antikörpern zu ermöglichen, die schlecht an Protein A binden. Die anderen IgG-Isotypen binden entweder an Protein A oder an Protein G.
Sollte ich heterologe Spike-in-DNA für die Quantifizierung verwenden?
Unser Protokoll basiert weitgehend auf dem verbesserten CUT&RUN-Protokoll. Hier zeigen die Autoren, dass eine genaue Quantifizierung unter Verwendung heterologer Spike-in-DNA oder Carry-over-E.-coli-DNA aus der pAG-MNase-Reinigung möglich ist. Daher ist die Zugabe von heterologer Spike-in-DNA nicht erforderlich.
Ist es möglich, die Zellen vor der Immobilisierung zu fixieren?
Es ist möglich, Ihre Proben zu fixieren, um z. B. die Dissoziation größerer Proteinkomplexe von der DNA im Verlauf des Experiments zu vermeiden. Sie können entweder Ihr bewährtes Vernetzungsverfahren anwenden oder milde Vernetzungsbedingungen unter Verwendung von Formaldehyd in einer niedrigeren Konzentration von 0,1 % verwenden. Die Vernetzung mit 1 % Formaldehyd kann das Signal tatsächlich verringern, was möglicherweise auf die Maskierung von Epitopen zurückzuführen ist. In diesen Fällen ist eine niedrigere Konzentration des Vernetzers vorzuziehen.
Ist es möglich, die CUT&RUN-Antikörper-Online-Produktsets mit pflanzlichen Gewebeproben zu verwenden?
Die CUT&RUN-Methode kann auf pflanzliche Gewebeproben angewendet werden. Ein wesentlicher Schritt zusätzlich zu den im Protokoll aufgeführten ist die Erzeugung von Sphäroblasten, damit die Plasmamembran für die Applikation der Antikörper und des MNase-Fusionsproteins permeabilisiert werden kann. Alternativ können Sie auch isolierte Zellkerne als Probenmaterial verwenden.
Der CUT&RUN-Kaninchen-Anti-h4K27me3-Antikörper als Positivkontrolle ( ABIN6923144) und der CUT&RUN-Meerschweinchen-Anti-Kaninchen-IgG-Antikörper als Negativkontrolle ( ABIN101961) sowie die ConA-Beads ( ABIN6923139 oder ABIN6952467) sind für die Verwendung mit Pflanzenproben geeignet.
Ist es möglich, CUT&RUN an RNA-bindende Proteine anzupassen?
Die für CUT&RUN verwendete MNase akzeptiert auch RNA als Substrat, so dass es möglich sein könnte, das CUT&RUN-Protokoll für die Verwendung mit RNA-bindenden Proteinen anzupassen.
RNA im Zytoplasma wird von der Abbaumaschinerie angezogen, wenn ihr die 5'-Kappe und der 3'-Poly-A-Schwanz fehlen. Daher würde ich vorschlagen, mit isolierten Zellkernen zu arbeiten. Dies hat den zusätzlichen Vorteil, dass man das Digitonin in den Puffern weglassen kann, das als Cholesterinersatz und zur Permeabilisierung der Zellmembran verwendet wird. Die isolierten Zellkerne können dann mit magnetischen ConA-Beads wie bei einem CUT&RUN-Experiment immobilisiert werden. Ein Antikörper gegen das interessierende Protein wird hinzugefügt, und anschließend wird die pAG-MNase an den Antikörper gebunden, wodurch die MNase in die Nähe der interessierenden RNA gebracht wird. Die isolierte RNA kann dann durch reverse Transkription in cDNA umgeschrieben werden, um Ihre Sequenzierungsbibliothek zu erstellen.
Um jegliche kontaminierte DNA zu beseitigen, sollten Sie nach der RNA-Prep und vor der Bibliotheksvorbereitung eine DNase-Behandlung in das Protokoll aufnehmen. Eine Normalisierung auf der Grundlage der mit der MNase übertragenen E. coli-DNA oder einer Spike-in-DNA ist in diesem Fall keine Option. Ziehen Sie stattdessen in Erwägung, die Gesamtlesezahlen zur Normalisierung zwischen den Proben zu verwenden oder eine Referenz-RNA mit einer bekannten Konzentration einzubeziehen.
Kann ich anstelle des Proteinase-K-Verdaus die Proteine in den CUT&RUN-Produktkomplexen durch Hitze denaturieren?
Wir raten von dieser Möglichkeit ab: Die interessierende DNA liegt zu diesem Zeitpunkt in einem Komplex aus der DNA, dem Antigen, dem entsprechenden Antikörper und der pAG-MNase vor. Durch das Kochen dieses Komplexes wird die DNA wahrscheinlich zusammen mit denaturiertem Protein ausgefällt. Dies betrifft auch in erster Linie die kurzen CUT&RUN-Produkte und nicht die größeren DNA-Moleküle, was zu einem geringeren Signal-Rausch-Verhältnis in Ihrer Bibliothek führt und möglicherweise auch die Komplexität der Bibliothek verringert. Dieser Effekt wird durch die niedrigere Schmelztemperatur dieser kurzen Moleküle im Vergleich zu den längeren kontaminierenden DNA-Molekülen noch verschärft.
Was ist bei der DNA-Extraktion vor der Bibliotheksvorbereitung vorzuziehen: Extraktion mit Phenol-Chloroform oder Affinitätsreinigung mit einer Säule?
Ein potenzielles Problem bei der Verwendung von SPRI-Beads für die Reinigung von DNA-Fragmenten ist die Verschleppung von aktiver Proteinase K, die die nachfolgende PCR-Amplifikation beeinträchtigen kann. Daher ist eine Phenol-Chloroform-Extraktion vorzuziehen, um eine vollständige Denaturierung der Proteinase K zu gewährleisten.
Ist es möglich, die CUT&RUN-Bibliotheken mit Single-End- statt mit Paired-End-Sequenzen zu sequenzieren?
Eine Single-End-Sequenzierung anstelle einer Paired-End-Sequenzierung ist möglich. Sie hat jedoch Nachteile gegenüber der Paired-End-Sequenzierung: (i) Bei häufig vorkommenden Zielmolekülen wie Histonmarkern oder Transkriptionsfaktoren wird eine große Anzahl von Bindungsstellen erwartet. Die Paired-End-Sequenzierung erleichtert die eindeutige Zuordnung zur richtigen genomischen Position. Diese zusätzliche Information verringert die erforderliche Sequenzierungstiefe. (ii) MNase verdaut die Ziel-DNA bis zu dem Abschnitt, der von dem interessierenden Protein bedeckt wird. Bei der Paired-End-Sequenzierung wird dieser Fußabdruck sichtbar, während die Information bei der Single-End-Sequenzierung verloren geht.
Targets in CUT&RUN / CUT&Tag Veröffentlichungen
Basierend auf veröffentlichten Arbeiten sind die Mehrzahl der für die mittels CUT&RUN und CUT&Tag gezielten Fragmentierung genomischer DNA genutzten Antigene Histonmodifikationen wie das für Repression typische H3K4me3 sowie H3K27me3, charakteristisch für geschlossenes Chromatin. Andere Proteinkandidaten sind Transkriptionsregulatoren wie die Transkriptionsfaktoren CTCF und SALL4, Chromatinmodifizierer wie HDAC2 oder in den Nukleinsäurestoffwechsel involvierte Enyzme wie Pol II. Die meisten CUT&RUN und CUT&Tag Studien nutzen bislang Proben menschlichen oder murinen Ursprungs. Für viele der untersuchten Antigene bieten wir bereits Antikörper an, die insbesondere für CUT&RUN und/oder CUT&Tag validiert sind.
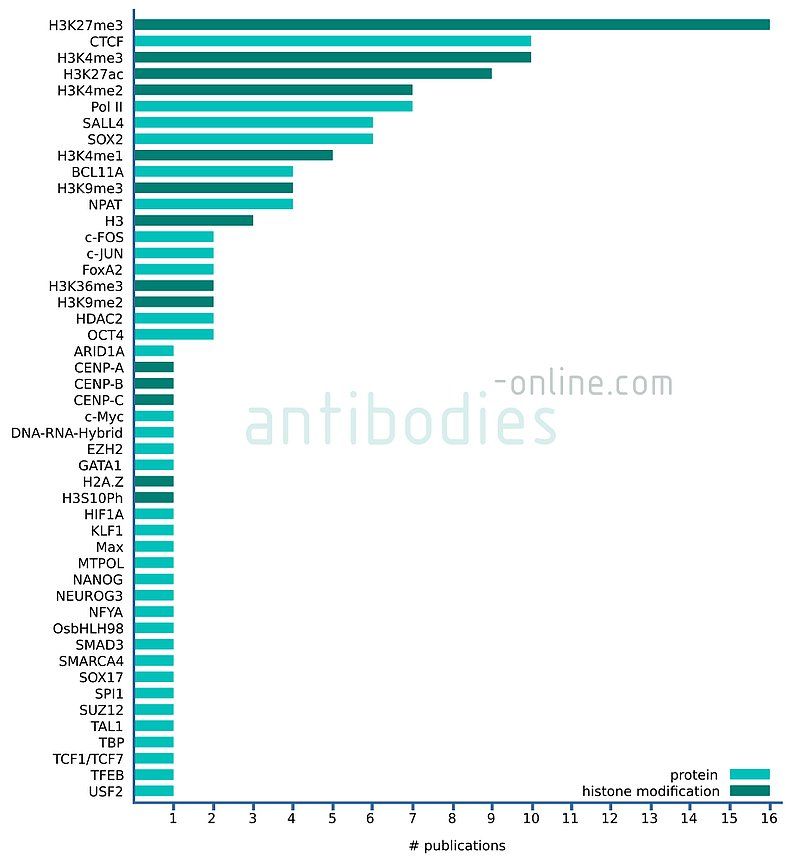
Abb. 1: Anzahl der Veröffentlichungen, in denen die angegebenen Antigene im Zusammenhang mit CUT&RUN und CUT&Tag erwähnt werden, basierend auf 45 Zeitschriftenartikeln, die von 2018 bis 2021 veröffentlicht wurden.
Zusätzliche Ressourcen zum Thema CUT&RUN
- antibodies-online CUT&RUN Protocol @ protocols.io
- CUT&RUN - Improved Method to Study Protein-DNA Interactions
- Discover our CUT&RUN Sets - Download Flyer
- CUT&Tag - Workflow, Advantages, Products
Referenzen
- : "An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites." in: eLife, Vol. 6, (2018) (PubMed).
- : "RSC-Associated Subnucleosomes Define MNase-Sensitive Promoters in Yeast." in: Molecular cell, Vol. 73, Issue 2, pp. 238-249.e3, (2019) (PubMed).
- : "Improved CUT&RUN chromatin profiling tools." in: eLife, Vol. 8, (2019) (PubMed).
- : "Efficient chromatin accessibility mapping in situ by nucleosome-tethered tagmentation." in: eLife, Vol. 9, (2020) (PubMed).





