CUT&Tag
CUT&Tag (Cleavage Under Targets and Tagmentation) bietet einen neuartigen Ansatz für die Epigenetik-Forschung. Die Methode ist darauf ausgelegt, genomweite Histonvarianten und posttranslationale Modifikationen, Transkriptionsfaktor-Bindungsstellen und Chromatin-assoziierte Komplexe zu kartieren.
CUT&Tag Arbeitsablauf

CUT&Tag wird in situ an immobilisierten, intakten Zellen ohne Quervernetzung durchgeführt. Die DNA-Fragmentierung wird durch die Tagmentierung genomischer DNA durch eine hyperaktive Tn5-Transposase erreicht, die mit Sequenzierungsadapter-Duplexen beladen und mit Protein A und/oder Protein G (pA/G-Tn5) fusioniert wird. Das Fusionsprotein wird über seine Protein A- und/oder Protein G-Anteile an einen Antikörper gegen das gewünschte Ziel gebunden und führt so zu einer Tagmentierung in der Nähe des Zielantigens. CUT&Tag-Produkte eignen sich ohne weitere Endpolishing-Schritte zur Bibliotheksgenerierung für Next Generation Sequencing (NGS).
Vorteile von CUT&Tag
- In situ auf nicht-fixierten Zellen durchgeführt; keine Chromatinfragmentierung notwendig.
- Niedriger Hintergrund und hohe Sensitivität erfordern eine geringe Sequenziertiefe.
- Keine End-Polishing- und Sequenzieradapter-Ligationsschritte notwendig.
- Möglich mit niedrigen Zellzahlen bis zu 100 Zellen, abhängig vom Antigen.
- Einfach, schnell, gut automatisierbar.
- Genaue Quantifizierung unter Verwendung von carry-over E. coli DNA aus der pAG-Tn5-Aufreinigung.
CUT&RUN + CUT&Tag Handbuch
Laden Sie kostenlos unser Handbuch zu CUT&RUN and CUT&Tag herunter!
Produkte für CUT&Tag
antibodies-online bietet flexible Lösungen für Ihre CUT&Tag-Experimente. Suchen Sie sich entweder Kontroll-Antikörper, Sekundär-Antikörper oder magnetische Beads aus unserem CUT&Tag-Produktangebot aus und kontaktieren Sie unser Team von Biologen, um ein fein abgestimmtes Set von Reagenzien zusammen mit einem optimierten Protokoll zu erhalten.
- CUT&Tag Sekundärantikörper
- Signalverstärkung in CUT&Tag
- Effiziente pAG-Tn5-Fusionsanbindung
- Optimiert für CUT&Tag Assays
- Bessere Handhabung im Vergleich zu Silica ConA Beads, vergleichbare Leistung
- Validiert für CUT&RUN und CUT&Tag
- Spezifischer Nachweis von Maus IgG (H&L)
- 3 CUT&Tag Zitationen verfügbar
Epigenetische Antikörper für CUT&Tag
antibodies-online bietet Antikörper gegen häufige Histonmodifikationen und Transkriptionsfaktoren an. Sie sind als Positivkontrolle geeignet und wichtig, um den Erfolg der CUT&Tag-Methode zu beurteilen.
Häufig gestellte Fragen
Wie wähle ich zwischen CUT&RUN und CUT&Tag?
Bei CUT&Tag werden die Sequenzierprimer an die gespaltene DNA angehängt und es ist kein zusätzlicher Annealing-Schritt notwendig. Es funktioniert gut für nukleosomale und eng gebundene Proteine. Die Methode ist auch weniger anfällig für Hintergrundsignale aufgrund von DNA-Schäden.
CUT&RUN hingegen ist für Transkriptionsfaktoren (Komplexe) oder weniger fest an DNA gebundene Proteine vorzuziehen. Es hat auch eine bessere Auflösung als CUT&Tag.
Ist es möglich, CUT&Tag mit pflanzlichen Gewebeproben zu verwenden?
CUT&Tag auf isolierten Pflanzenzellen wurde für die Profilierung von H3K4me3-Histonmarkierungen in verschiedenen Baumwollgewebetypen unter Verwendung isolierter Zellkerne beschrieben14. Unsere Positivkontrolle H3K27me3 H3K4me3-Antikörper und die Negativkontrolle Meerschweinchen-Anti-Kaninchen-IgG-Antikörper sowie die ConA-Beads sind für die Verwendung mit Pflanzenproben geeignet.
Kann ich die Proteine im CUT&Tag-Produktkomplex anstelle einer Proteinase-K-Behandlung denaturieren?
Wir raten von dieser Möglichkeit ab: Die interessierende DNA liegt zu diesem Zeitpunkt in einem Komplex vor, der aus der DNA, dem interessierenden Protein, dem entsprechenden Antikörper und dem pA/G-Tn5 besteht. Ein Abkochen dieses Komplexes wird wahrscheinlich die DNA zusammen mit denaturiertem Protein ausfällen. Dies wird auch in erster Linie die kurzen CUT&Tag-Produkte betreffen und nicht größere DNA-Moleküle, was zu einem verringerten Signal-Rausch-Verhältnis in Ihrer Bibliothek führt und möglicherweise auch die Komplexität der Bibliothek verringert. Dieser Effekt wird durch die niedrigere Schmelztemperatur dieser kurzen Moleküle im Vergleich zu den längeren kontaminierenden DNA-Molekülen noch verstärkt.
Welche sekundären Antikörper kann ich für CUT&Tag verwenden?
- (105)
- (3)
- (2)
Kann ich AMPure CP-Beads zur Aufreinigung von Tagmentierungsprodukten anstelle der Phenol-Chloroform-Extraktion verwenden?
In der Originalpublikation, die die CUT&Tag-Methode beschreibt, erwähnen die Autoren die Verwendung von AMPure XP-Beads für die Aufreinigung der DNA nach der Tagmentierung und dem Proteinase-K-Verdau1. Ein mögliches Problem ist die Verschleppung von aktiver Proteinase K, die die nachfolgende PCR-Amplifikation stören kann. Daher empfehlen die Autoren nun die Phenol-Chloroform-Extraktion, um eine vollständige Denaturierung der Proteinase K zu gewährleisten15.
Warum ist die DNA-Ausbeute so gering?
CUT&Tag wird mit geringen Zellzahlen durchgeführt und das Hintergrundsignal ist deutlich geringer als z.B. bei ChIP. Aufgrund dieser beiden Faktoren ist die Menge der zurückgewonnenen DNA oft zu gering, um mit einem fluorometrischen Assay oder durch Kapillarelektrophorese zuverlässig gemessen zu werden. Die PCR-Amplifikation von kleinen CUT&RUN-Produkten, d. h. weniger als 50 bp, kann problematisch sein und kommt daher nicht in Frage.
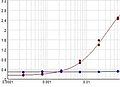
Um den Erfolg der CUT&Tag-Methode zu beurteilen, sollten (eine) positive Kontrollprobe(n) mit dem Positivkontroll-H3K4m3-Antikörper ABIN3023254 und/oder dem Positivkontroll-H3K27me3-Antikörper ABIN6923144 eingesetzt werden. Die mit diesen Antikörpern präparierten DNA-Fragmente können mittels sensitiver Elektrophorese auf einem Bioanalyzer oder Tapestation oder fluorometrisch auf einem Qubit- oder Nanodrop-Fluorometer gemessen werden. Bei Verwendung der CUT&RUN-Positivkontrolle ABIN6923144 (roter Punkt) (oder eines anderen für nukleosomale Marker spezifischen Antikörpers) sollte in der Kapillarelektrophorese eine Leiter sichtbar sein, die einem Vielfachen der 147 bp langen Nukleosomen entspricht.
Verwandte Informationen und Produkte
CUT&RUN - Spaltung unter Targets und Freigabe mittels Nuklease: Informationen und Produkte
Referenzen
: "Efficient chromatin profiling of H3K4me3 modification in cotton using CUT&Tag." in: Plant methods, Vol. 16, pp. 120, (2020) (PubMed).: "Improved CUT&RUN chromatin profiling tools." in: eLife, Vol. 8, (2019) (PubMed).
: "The three-dimensional structure of a Tn5 transposase-related protein determined to 2.9-A resolution." in: The Journal of biological chemistry, Vol. 274, Issue 17, pp. 11904-13, (1999) (PubMed).
: "ChIC and ChEC; genomic mapping of chromatin proteins." in: Molecular cell, Vol. 16, Issue 1, pp. 147-57, (2004) (PubMed).
: "Multiplex single cell profiling of chromatin accessibility by combinatorial cellular indexing." in: Science (New York, N.Y.), Vol. 348, Issue 6237, pp. 910-4, (2015) (PubMed).
: "An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites." in: eLife, Vol. 6, (2018) (PubMed).
: "CUT&Tag for efficient epigenomic profiling of small samples and single cells." in: Nature communications, Vol. 10, Issue 1, pp. 1930, (2019) (PubMed).
Henikoff, S. & Henikoff, J. G. Profiling the epigenome at home. bioRxiv 1–18 (2020). doi:10.1101/2020.04.15.043083
Bartosovic, M., Kabbe, M. & Castelo-Branco, G. Single-cell profiling of histone modifications in the mouse brain. bioRxiv 2020.09.02.279703 (2020). doi:10.1101/2020.09.02.279703
Wu, S. J. et al. Single-cell analysis of chromatin silencing programs in developmental and tumor progression. bioRxiv 2020.09.04.282418 (2020). doi:10.1101/2020.09.04.282418
Kaya-Okur, H., Henikoff, S. & Henikoff, S. Bench top CUT&Tag. protocols.io 1–19 (2019). Available at: https://www.protocols.io/view/bench-top-cut-amp-tag-z6hf9b6.




