Das Coronavirus 2 des Schweren Akuten Respiratorischen Syndroms (SARS-CoV-2) gehört zu den umhüllten positiv-sensitiven RNA-Viren. Dieses Virus zeichnet sich durch keulenförmige Stacheln auf der Oberfläche und eine einzigartige Replikationsstrategie aus. Der Zelleintritt von Coronaviren hängt von der Bindung der viralen Spike-Proteine (S-Proteine) an zelluläre Rezeptoren und vom Priming des S-Proteins durch Proteasen der Wirtszelle ab. Die Klärung der Frage, welche zellulären Faktoren von SARS-CoV-2 für den Eintritt in die Zelle genutzt werden, könnte Einblicke in die virale Übertragung gewähren und therapeutische Ziele aufzeigen.1
Im Folgenden werden der Replikationszyklus von SARS-CoV-2 sowie mögliche Hemmstoffe und ihre jeweiligen Angriffspunkte erläutert.Diese Zusammenstellung basiert auf der aktuellen Literatur.
Stadien des Lebenszyklus von SARS-CoV-2:
SARS-CoV-2 Replikationszyklus
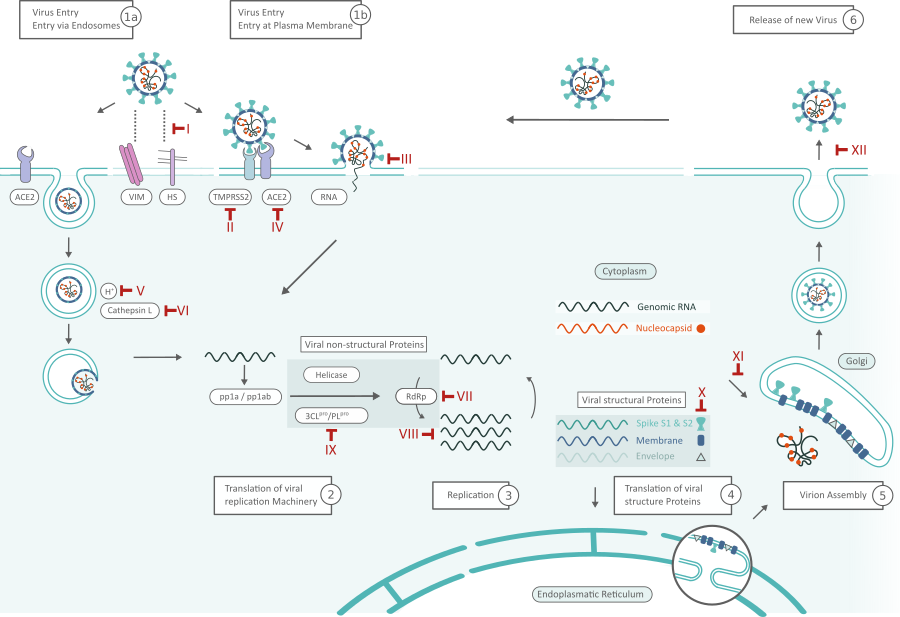
SARS-Cov-2 Replikationszyklus und Inhibitoren. Mögliche Ziele für Inhibitoren sind rot markiert und mit römischen Ziffern nummeriert. Klicken Sie hier für eine größere Ansicht
Viruseintrag (1)
SARS-CoV-2 kann auf zwei Wegen in die Zelle eindringen, entweder über Endosomen oder über eine Plasmamembranfusion. (Auf beiden Wegen) Spike-Proteine (S1, S2) von SARS-CoV-2 vermitteln die Anheftung an die Membran einer Wirtszelle und binden Angiotensin-konvertierendes Enzym 2 (ACE2) als Eintrittsrezeptor.1 Inhibitoren wie Griffithsin (Inhibitor III) binden an das Spike-Glykoprotein und verhindern so den Eintritt des Virus. Zelloberflächen-Vimentin (VIM) fungiert als kritischer Co-Rezeptor und ist für die erfolgreiche Bindung von ACE-2 unerlässlich.2 Die Bindung von Heparansulfat (HS) an die Rezeptorbindungsdomäne (RBD) verstärkt ebenfalls die Bindung an ACE2. Die virale Adhäsion kann durch exogenes Heparin gehemmt werden. Heparin konkurriert mit HS um die Bindung des SARS-CoV-2 S-Proteins.3
Wenn die Virionen in die Endosomen aufgenommen werden, aktiviert Cathepsin L das Spike-Protein. Die pH-abhängige Cysteinprotease kann durch lysosomotrope Wirkstoffe, wie Bafilomycin A1 oder Ammoniumchlorid, blockiert werden. (Inhibitorklassen IV,V) blockiert werden. Alternativ kann das Spike-Protein zwischen der S1- und S2-Domäne durch die zelluläre Serinprotease TMPRSS2 in unmittelbarer Nähe des ACE2-Rezeptors gespalten werden, wodurch die Fusion der viralen Membran mit der Plasmamembran eingeleitet wird (Inhibitor II: Camostat). 1 Der Eintritt in die Plasmamembran löst weniger wahrscheinlich eine antivirale Immunität der Wirtszelle aus und ist daher effizienter für die virale Replikation. 4
SARS-CoV-2 Inhibitor Screening Kit
Dieses Inhibitor-Screening-ELISA-Paar wurde entwickelt, um die Identifizierung und Charakterisierung von SARS-CoV-2-Inhibitoren zu erleichtern.
Produktdetails anzeigenVerwandte Produkte: SARS-CoV-2-Spike-Proteine | SARS-CoV-2 neutralisierende Antikörper | SARS-CoV-2 S1-Protein Mutationen | ACE2-Antikörper | TMPRSS2-Antikörper
- (4)
- (5)
- (1)
- (1)
- (2)
- (3)
- (1)
Übersetzung der viralen Replikationsmaschinerie (2) und Replikation (3)
Nachdem die virale RNA in die Wirtszelle freigesetzt wurde, werden Polyproteine translatiert. Die genomische RNA des Coronavirus kodiert für nicht-strukturelle Proteine (NSPs), die eine entscheidende Rolle bei der viralen RNA-Synthese spielen, sowie für Strukturproteine, die für den Zusammenbau des Virions wichtig sind. Zunächst werden die Polyproteine pp1a und pp1ab translatiert, die von der Papain-ähnlichen Protease (PLpro, Nsp3) und der 3C-ähnlichen Protease (3CLpro, Nsp5) (Inhibitor VIII) gespalten werden, um funktionale NSPs wie Helicase oder den RNA-Replicase-Transkriptase-Komplex (RdRp) zu bilden. 5 RdRp kann insbesondere durch Virostatika wie Favipiravir oder Penciclovir (Inhibitor VI) gehemmt werden; die Replikation viraler RNA im Allgemeinen durch Kinase-Signalweg-Inhibitoren wie Saracatinib (Inhibitor VII). Das Expressionsniveau des N-Proteins kann durch Resveratrol (Inhibitor X) verringert werden.7
Eines der ersten translatierten Proteine ist der Wirtsabschaltfaktor Nsp1. Dieses virale Protein stört die Translation und bewirkt einen beschleunigten Abbau der Wirts-mRNA und unterdrückt so die angeborene Immunantwort des Wirts. 8
Verwandte Produkte: SARS-CoV-2 Non-Structural Proteins | SARS-CoV-2 N-Proteine | SARS-CoV-2 N-Antikörper
SARS-CoV-2 S-Mutationsproteine
Wir unterstützen Ihre Forschung mit zuverlässigen SARS-CoV-2 Mutationsproteinen. B.1.1.7 / P.1 / B.1.351 / S Protein Wildtyp. Mehr als 400 Labore in Diagnostik, Pharma und Wissenschaft haben unsere SARS-CoV-2-Forschungstools verwendet.
Erforschen Sie unsere Vielfalt an Mutationsproteinen- (9)
- (8)
- (1)
- (2)
- (3)
- (2)
Translation viraler Strukturproteine (4) und Zusammenbau des Virions (5)
RdRp (Nsp12) ist für die Replikation der Strukturprotein-RNA verantwortlich. Die Strukturproteine S, Envelope (E), Membrane (M) werden von Ribosomen übersetzt, die an das endoplasmatische Retikulum (ER) gebunden sind. Das ER bildet Doppelmembranbläschen (DMVs), in denen die virale RNA repliziert und vor dem angeborenen Immunsystem des Wirts geschützt wird. Nsp3 erzeugt Poren, durch die die virale RNA die DMVs verlässt, um das Virion zu bilden. Die Nukleokapsidproteine (N) verbleiben im Zytoplasma und werden aus genomischer RNA zusammengesetzt. Sie verschmelzen mit dem Virion-Vorläufer, der dann vom ER durch den Golgi-Apparat über kleine Vesikel zur Zelloberfläche transportiert wird.
Verwandte Produkte: SARS-CoV-2-Spike-Proteine | SARS-CoV-2 S1-Protein Mutationen | SARS-CoV-2 S-Antikörper
- (8)
- (15)
- (1)
- (5)
- (3)
- (1)
- (2)
Freisetzung des Virus (6)
Die Virionen werden dann durch Exozytose aus der infizierten Zelle freigesetzt und suchen eine andere Wirtszelle auf. Oseltamivir hemmt die Abspaltung von Sialinsäuren durch Neuroamidase von den Zellrezeptoren und verhindert so die Freisetzung neu gebildeter Virionen von der Zelloberfläche (Inhibitor XI). Ein Merkmal, das SARS-CoV-2 von anderen Coranaviren wie z. B. SARS-CoV-2 unterscheidet, ist eine zweite Spaltstelle im S-Protein. Es wird angenommen, dass die proteolytische Spaltung dieser Furin-Spaltstelle während des Zusammenbaus des Virions das Virus für den Eintritt in die Wirtszellen vorbereitet. Bestimmte Mutationen an dieser Stelle sind auch charakteristisch für die SARS-CoV-2-Varianten der Gruppen alpha (B.1.1.7), beta (B.1.351) und delta (B.1.617.2). 8
Brauchen Sie Hilfe? Rufen Sie unseren PhD-Kundenservice an!
- Wir helfen Ihnen bei der Suche nach dem richtigen Produkt für Ihre Forschung.
- Wir bieten zuverlässige Antikörper, Kits, Proteine und Lysate für die COVID-19 Forschung.
- Kontaktieren Sie uns per E-Mail oder Telefon: (877) 302 8632 (US) oder +49 241 95 163 153 (International)
Verwandte Informationen und Produkte
SARS-CoV-2 neutralisierende Antikörper auf Basis von CR3022
Mutationen von SARS-CoV-2 S-Protein
SARS-CoV Protein Interactom / Poster und Produkte
SARS-CoV-2 RT-PCR Kits und qPCR Kits
Referenzen
- (1) Hoffmann et al.: SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor, Cell (2020), PDF
- Suprewicz et al.: Vimentin binds to SARS-CoV-2 spike protein and antibodies targeting extracellular vimentin block in vitro uptake of SARS-CoV-2 virus-like particles, BioRxiv preprint (2021), [DOI]
- (2) Henderson et al: Controlling the SARS-CoV-2 Spike Glycoprotein Conformation, Nat Struct Mol Biol. (2021), [ DOI]
- (3) Shirato, K., Kawase, M. & Matsuyama, S.: Wild-type human coronaviruses prefer cell-surface TMPRSS2 to endosomal cathepsins for cell entry, Virology 517, 9–15 (2018), [DOI]
- (4) Zhavoronkov et al.: Potential COVID-2019 3C-like Protease Inhibitors Designed Using Generative Deep Learning Approaches, ChemRxiv (2020), [DOI]
- (5) Shin et al.: Saracatinib Inhibits Middle East Respiratory Syndrome-Coronavirus Replication In Vitro, Viruses, 10(6):283 (2018), [DOI]
- (6) Lin, S. C., Ho, C. T., Chuo, W. H., Li, S., Wang, T. T., & Lin, C. C. : Effective inhibition of MERS-CoV infection by resveratrol, BMC Infectious Diseases, 17(1)(2017), [DOI]
- (7) McKimm‐Breschkin: Influenza neuraminidase inhibitors: Antiviral action and mechanisms of resistance. Influenza and Other Respiratory Viruses 7(Suppl. 1), 25–36., [PMC]
- (8) Scudellari M.: How the coronavirus infects cells — and why Delta is so dangerous, Nature (2021), [DOI]

Creative mind of antibodies-online with a keen eye for details. Proficient in the field of life-science with a passion for plant biotechnology and clinical study design. Responsible for illustrated and written content at antibodies-online as well as supervision of the antibodies-online scholarship program.
Mehr Informationen zum Autor



