Histone Code
Kategorien
Histone assoziieren mit der DNA und bilden Nukleosomen, die grundlegende Struktureinheit der DNA in Eukaryoten. Sie bestehen aus acht Histonen, jeweils zwei Kopien der Histonproteine H2A, H2B, H3 und H4 eingewickelt in DNA. Der "Histon-Code" ist eine Hypothese, die besagt, dass die DNA-Transkription weitgehend durch posttranslationale Modifikationen (PTM) an diesen Histonproteinen reguliert wird. Zusammen mit ähnlichen Modifikationen wie der DNA-Methylierung ist er Teil des epigenetischen Codes.
PTMs wie Methylierung oder Acetylierung dienen als epigenetisches Markierungssystem, das andere Proteine rekrutiert, indem es das modifizierte Histon über dafür spezialisierte Proteindomänen spezifisch erkennt und nicht einfach die Interaktion zwischen Histon und der darunter liegenden DNA stabilisiert oder destabilisiert. Diese rekrutierten Proteine wirken dann, um die Chromatinstruktur aktiv zu verändern und die Transkription zu fördern oder zu unterdrücken.
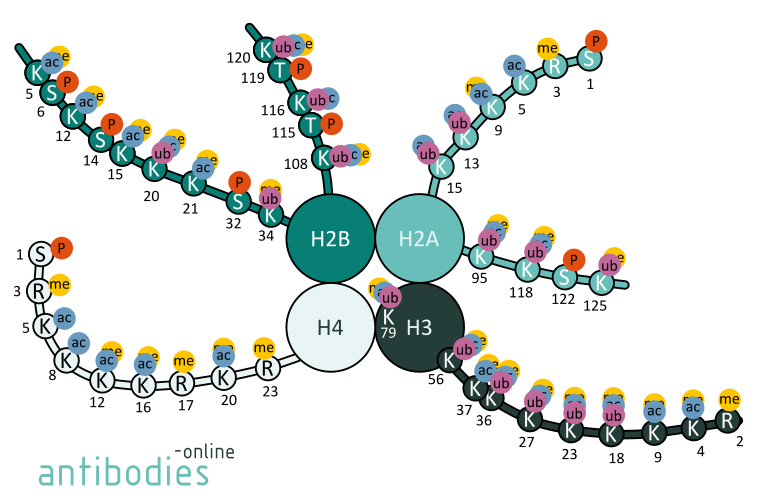
Fig. 1: Einige Histonmodifikationen an ausgesuchten Aminosäuren der Histone H2A, H2B, H3, and H4.
Histon-Modifikationen und ihre Auswirkungen
Eine Histonmodifikation ist eine kovalente posttranslationale Modifikation (PTM) an Histonproteinen. Histon-Modifikationen wirken bei verschiedenen biologischen Prozessen wie der Transkriptionsaktivierung/-inaktivierung, der Chromosomenverpackung und der DNA-Schädigung/Reparatur.
Epigenetische Veränderungen bieten die einzigartige Möglichkeit, die Genexpression auf reversible Weise zu verändern - ein Einfallstor für neue Therapieansätze. Das Verständnis der Veränderungen hat das Potenzial, die Mechanismen des Alterns, der menschlichen Entwicklung und die Entstehung verschiedener Gesundheitszustände, z. B. Krebs, Herzerkrankungen und psychische Erkrankungen, zu erklären.
Histon-Acetylierung
Generell bestimmt die Histon-Acetylierung den Histonaufbau sowie die Faltung und Kompaktheit der DNA-Histon-Interaktion und stellt somit einen Schalter zwischen kondensierter und unkondensierter Chromatinstruktur dar. Letztere, bekannt als Euchromatin, ist transkriptionell aktiv, während erstere, bekannt als Heterochromatin, transkriptionell inaktiv ist.
Histon-Acetyltransferasen (HAT) übertragen eine Acetylgruppe auf spezifische Lysinreste auf Histonen, normalerweise Reste im N-terminalen Schwanz. Acetylierte Lysin-Seitenketten der Histone verlieren ihre positive Ladung und damit die Fähigkeit, Salzbrücken mit dem negativ geladenen Phosphatgerüst der DNA zu bilden. H3- und H4-Histonproteine sind die primären Ziele von HATs.
Acetylation of H3K56, H3K64 und H3K122 oder H4K16 unterbricht die Chromatinstruktur und führt zu leicht gepacktem Euchromatin. Euchromatin ist an der aktiven Transkription der DNA beteiligt. Die entfaltete Struktur ermöglicht genregulatorischen Proteinen und RNA-Polymerase-Komplexen die Bindung an die DNA-Sequenz und führt so zu einer Steigerung der Gentranskription.
Histon H3K27m3 - Abschaltung der Transkription
H3K27m3 ist ein intensiv untersuchtes Ziel von epigenetischen Forschern auf der Suche nach inaktiven Genen. Im Gegensatz zu anderen Histon-Methylierungen gibt es für H3K27m3 nur eine bekannte Methyltransferase: EZH2. EZH2 ist für die Repression vieler Gene verantwortlich, die an der Entwicklung und Zelldifferenzierung beteiligt sind. Daher ist H3K27m3 entscheidend für die Repression von Entwicklungsgenen, die im Gegensatz zu H3K4m3. Wenn H3K27 trimethyliert ist, ist es eng mit inaktiven Genpromotoren assoziiert.
DNA- und Histon-Lysin-Methylierungssysteme sind hochgradig miteinander verknüpft und hängen für eine normale Chromatinfunktion in vivo mechanistisch voneinander ab. Nicht-methylierte DNA in CpG-Inseln kann als Teil einer genomischen Signatur fungieren, um H3K4- und H3K27-Trimethylierung zu rekrutieren und H3K36-Methylierung auszuschließen, wodurch möglicherweise Chromatin-Umgebungen geschaffen werden, die einzigartig für genregulatorische Elemente sind und die Transkriptionszustände modulieren können. Funktionelle Verbindungen zwischen 5-Hydroxymethylcytosin und Histon-Lysin-Methylierung stehen im Fokus aktueller Forschung, zum Beispiel die Fähigkeit von Uhrf1 und MeCP2, ebenfalls an 5hmC zu binden.
Die Mono- und Di-Methylierungszustände sind weniger untersucht; H3K27m2 zeigt jedoch eine ähnliche Verteilung wie H3K27, während H3K27m1 mit aktiven Promotoren assoziiert ist. H3K27 kann auch Ziel der Acetylierung sein. Lysinreste können nur methyliert oder acetyliert werden, wobei die Acetylierung zu entgegengesetzten Effekten führt - mit aktiver Transkription und Antagonismus von H3K27m3 regulierten Genen.
Histonmethylierung als therapeutisches Ziel
Eine Fehlregulation der Histon-Lysin-Methylierung wird mit verschiedenen Krebsarten und Entwicklungsstörungen in Verbindung gebracht. Daher wurde die Histon-Lysin-Methylierung als potenzielles therapeutisches Ziel angesehen, und klinische Studien mit verschiedenen Inhibitoren dieses Prozesses haben vielversprechende Ergebnisse gezeigt. Ein detaillierteres Verständnis der Histon-Lysin-Methylierung ist für die Aufklärung komplexer biologischer Prozesse und letztlich für die Entwicklung und Verbesserung von Krankheitsbehandlungen notwendig. Es gibt mehrere Ansätze zur Untersuchung von Histonen, darunter die Chromatin-Immunpräzipitation (zusammen mit ihren groß angelegten Varianten ChIP-on-chip und ChIP-Seq), die fluoreszierende In-situ-Hybridisierung oder die Bisulfit-Sequenzierung. In jüngster Zeit gewinnt die Spaltung unter Verwendung von Nuklease (CUT&RUN) als konsequente Verbesserung der ChIP-basierten Methoden immer mehr an Zugkraft.
antibodies-online bietet Ihnen ein breites Spektrum an Antikörpern für Ihre epigenetische Forschung. Durchstöbern Sie unser Portfolio weiter unten! Bei Fragen steht Ihnen unser Team von Biologen jederzeit per Chat, Kontaktformular oder E-Mail zur Verfügung.
Referenzen
: "The language of covalent histone modifications." in: Nature, Vol. 403, Issue 6765, pp. 41-5, (2000) (PubMed).: "Understanding the relationship between DNA methylation and histone lysine methylation." in: Biochimica et biophysica acta, Vol. 1839, Issue 12, pp. 1362-72, (2015) (PubMed).
: "Histone methylation and the DNA damage response." in: Mutation research. Reviews in mutation research, Vol. 780, pp. 37-47, (2020) (PubMed).
: "Independent manipulation of histone H3 modifications in individual nucleosomes reveals the contributions of sister histones to transcription." in: eLife, Vol. 6, (2018) (PubMed).




